1. 서 론
무각 와편모조류인 Cochlodinium polykrikoides Margalef는 1995년 이후 지속적으로 남해안에서 대규모 적조를 발생시키고 있다(Park et al., 2013; Lee et al., 2013). 주로 어류 폐사를 일으키는 유해 적조종으로 동아시아, 동남아시아, 북미, 유럽 등 전 세계적으로 넓게 분포하고 있다(Kudela and Gobler, 2012). C. polykrikoides는 large-subunit (LSU) ribosomal RNA gene을 이용하여 4가지 유전형으로 구분되며(Iwataki et al., 2008; Reñé et al., 2013), 동아시아 타입, 지중해 타입, 아메리카/말레이시아 타입, 필리핀 타입(각각 그룹Ⅰ,Ⅱ,Ⅲ,Ⅳ)으로 나뉜다. 필리핀 타입은 처음 필리핀해역에서 발견되었지만, 이후 동아시아 및 유럽에서도 발견이 되고 있어 4가지 유전형이 특정 해역에만 국한되어 분포하지는 않는다(Reñé et al., 2013; Park et al., 2014). 우리나라의 경우 동아시아타입(그룹Ⅰ)과 필리핀 타입(그룹Ⅳ)이 남해안과 대마난류 유입경로에서 발견되었다(Park et al., 2014; Park et al., 2018). Microsatellites 분자기법을 이용한 개체군 분석결과 우리나라와 일본해역에 3가지 유전형의 C. polykrikoides 개체군이 분포한다고 보고되었다(Nagai et al., 2009). 이러한 종내 다양한 유전형의 출현은 다양한 해양환경에 분포를 가능하게 하여 지속적인 적조발생의 원인이 될 수 있다(Park et al., 2014, Park et al., 2018; Kim et al., 2018).
Quantitative real-time PCR (qPCR)은 현장의 적조생물을 특이적으로 정량 검출하는 목적으로 널리 쓰이고 있다(e.g. Park et al., 2007, e.g. Park et al., 2016, e.g. Park et al., 2018; Lee et al., 2017; Kim et al., 2018). qPCR 기법 중 TaqMan probe를 이용하는 방법과 EvaGreen 시약을 이용하는 방법이 적조생물에 적용되고 있는데 2가지 방법 모두 저밀도로 분포하는 적조생물 검출에 유용하다(e.g. Park et al., 2014, e.g. Park et al., 2016, e.g. Park et al., 2018). 현재 3가지 유전형(그룹Ⅰ,Ⅲ,Ⅳ)의 C. polykrikoides을 식별할 수 있는 qPCR이 개발되어 있다(Park et al., 2014; Park et al., 2016). 본 연구에서는 어떤 유전형의 C. polykrikoides이 남해안에 분포하는지를 3년에(2014∼2016년) 걸쳐 조사하였다. 이를 위해 이전 연구에서 개발된 qPCR 기법을 이용하여 남해안 C. polykrikoides의 동아시아 타입과 필리핀 타입 출현 양상을 조사하였다. 조사범위는 통영∼완도해역을 대상으로 2가지 유전형의 출현 시기 및 출현 비율을 조사하였으며, 대마난류 유입세기와 필리핀 타입의 출현 시기를 비교하였다.
2. 재료 및 방법
2.1 배양주
통영해역에서 분리된 C. polykrikoides 단일 배양주(동아시아 타입)가 사용되었다. 동아시아 타입 C. polykrikoides 배양주는 f/2 배지(Guillard and Ryther, 1962)를 사용하여 20℃에서 형광등 빛으로 12:12시간 빛:암흑 주기 및 100 µmol photons‧m-1‧s-1의 광 밝기로 배양하였다. 1달간 배양된 세포는 원심분리하여 상등액을 제거하고 농축된 세포를 유전자 추출에 사용하였다. C. polykrikoides의 필리핀 타입은 배양주를 구하기 어려운 관계로 qPCR이 반응하는 영역을 합성하여 양성 대조구로 사용하였다. C. polykrikoides 필리핀 타입 특이적 qPCR 프라이머가(Park et al., 2014) 증폭하는 영역은(약 300 bp LSU rDNA) 마크로젠에 의뢰하여 합성하였다.
2.2 표층해수 채집
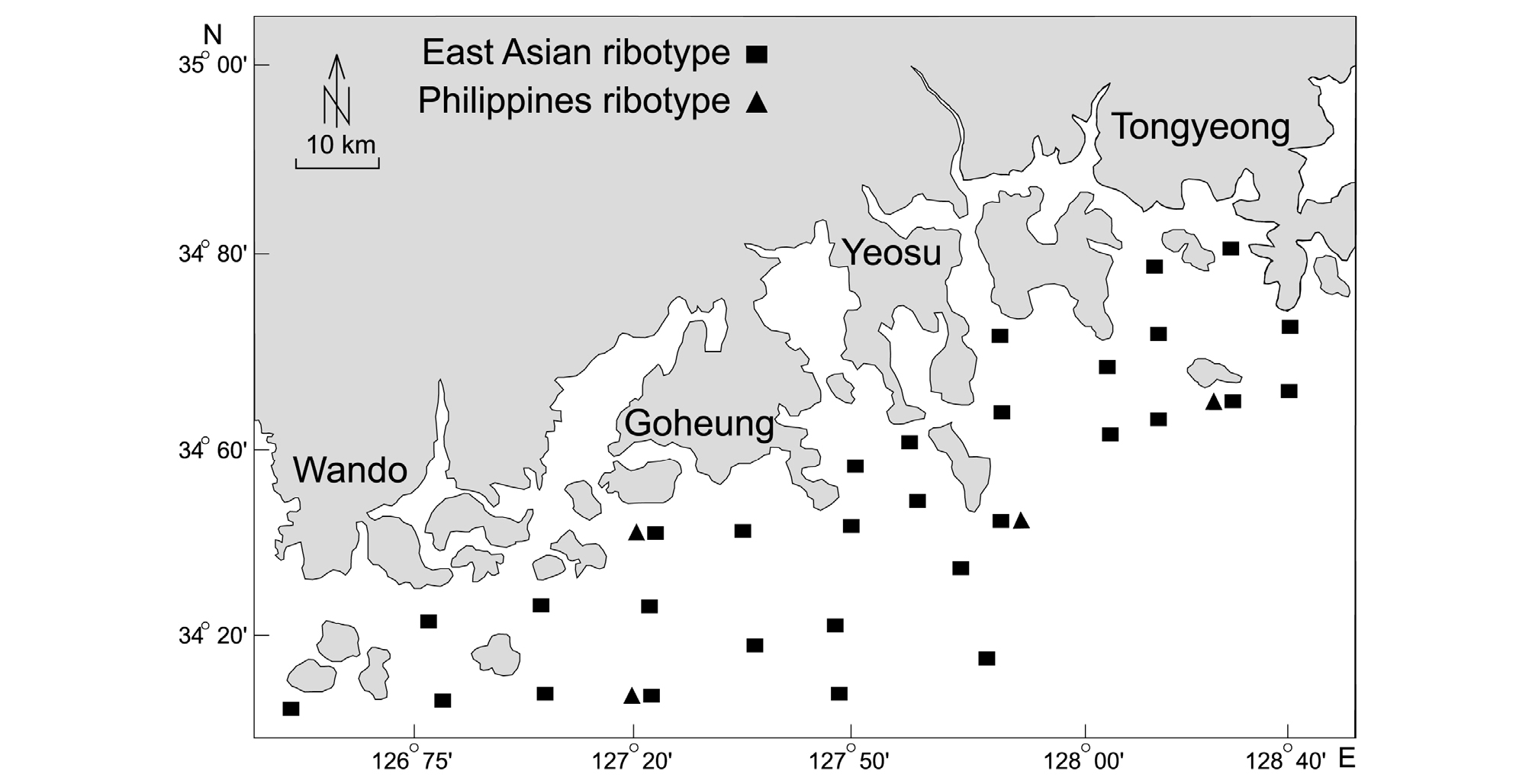
남해안 표층해수 시료는 국립수산과학원에서 수행하는 “적조광역조사”를 통해 채집되었다. 표층해수 시료는 2014∼2016년 6∼9월에 채집되었다(2016년은 6∼8월에 채집). 정점은 총 30개 정점을 대상으로 조사하였다(Fig. 1). 조사 횟수는 매주 조사를 하였으며, 경남해역 12개 정점은 주 1회 조사하였고, 전남해역 18개 정점은 매주 조사를 하되 홀수차는 18 정점, 짝수차는 8 정점을 조사하였다(Table 1). 따라서 매달 100개 해수시료를 채취하여 qPCR 분석을 실시하였다. 선박에서 표층해수 10L를 식물플랑크톤 넷(망구 10 um)으로 농축한 후 25 mm GF/C 필터지(Whatman, England)를 이용하여 여과한 후 DNA 추출 전까지 –20℃에 보관하였다.
Table 1. Sampling information for survey on distributions of two C. polykrikoides ribotypes in the South Sea Korea
| Number of sampling stations | Sampling period | Sampling frequency | |
| Odd-numbered week | Even-numbered week | ||
| 30 | 20 | June~September in 2014~2016 | Weekly |
2.3 DNA 추출
현장에서 표층해수를 여과하여 얻어진 GF/C 필터지는 DNeasy Blood & Tissue Kit (Qiagen, USA)을 이용하여 DNA를 추출하였다. 추출방법은 Qiagen 매뉴얼에 따라 추출하였다.
2.4 qPCR 분석
동아시아 타입을 위한 qPCR 분석시약은 TaqMan qPCR supermix (TaKaRa, Japan)를 사용하였다. 반응 전체량은 20 µL로 하였으며, 2 µL template DNA, 프라이머 최종 농도는 forward/reverse 각각 300 nM 및 TaqMan probe는 150 nM를 사용하였다(Table 2). qPCR 반응조건은 50℃ 2분, 95℃ 2분 후 40회 반복으로 95℃ 10초 그리고 60℃ 40초 반응시켰다. 필리핀 타입을 위한 real-time PCR 분석시약은 SsoFastTMEvaGreen® Supermix (Bio-Rad, USA)를 사용하였다. 반응 전체량은 20 µL로 하였으며, 2 µL template DNA, 프라이머 최종 농도는 forward/reverse 각각 300 nM를 사용하였다. qPCR 반응조건은 98℃ 2분 후 40회 반복으로 98℃ 5초 그리고 62℃ 10초 반응시켰다. 40회 반응 이후 melting curve 분석은 65∼95℃ 구간에서 0.2℃ 마다 10초간 실시하였다. 분석은 Rotor-Gene Q (Qiagen, USA)를 사용하였다.
Table 2. Sequence information for C. polykrikoides ribotype-specific qPCR
| Species | Direction | Name | Sequence (5'→3') | Reference |
| C. polykrikoides | Forward | PhiCPSF | TGCAAGTTTCAACCATCTCTCGC | Park et al., (2014) |
| Reverse | PhiCPSR | GAAAGCAAGTTCAATCGACGGTTT | ||
| Forward | CPLSUF | GCCGAGGATACCTGCAAAG | ||
| Reverse | CPLSUR | TGTCAGGACCCACGATCA | Park et al., (2016) | |
| Probe | CPLSUP | CTCACATGATCAGCGGCCGAGTACTAA |
2.5 qPCR 검출 양성반응 판정기준
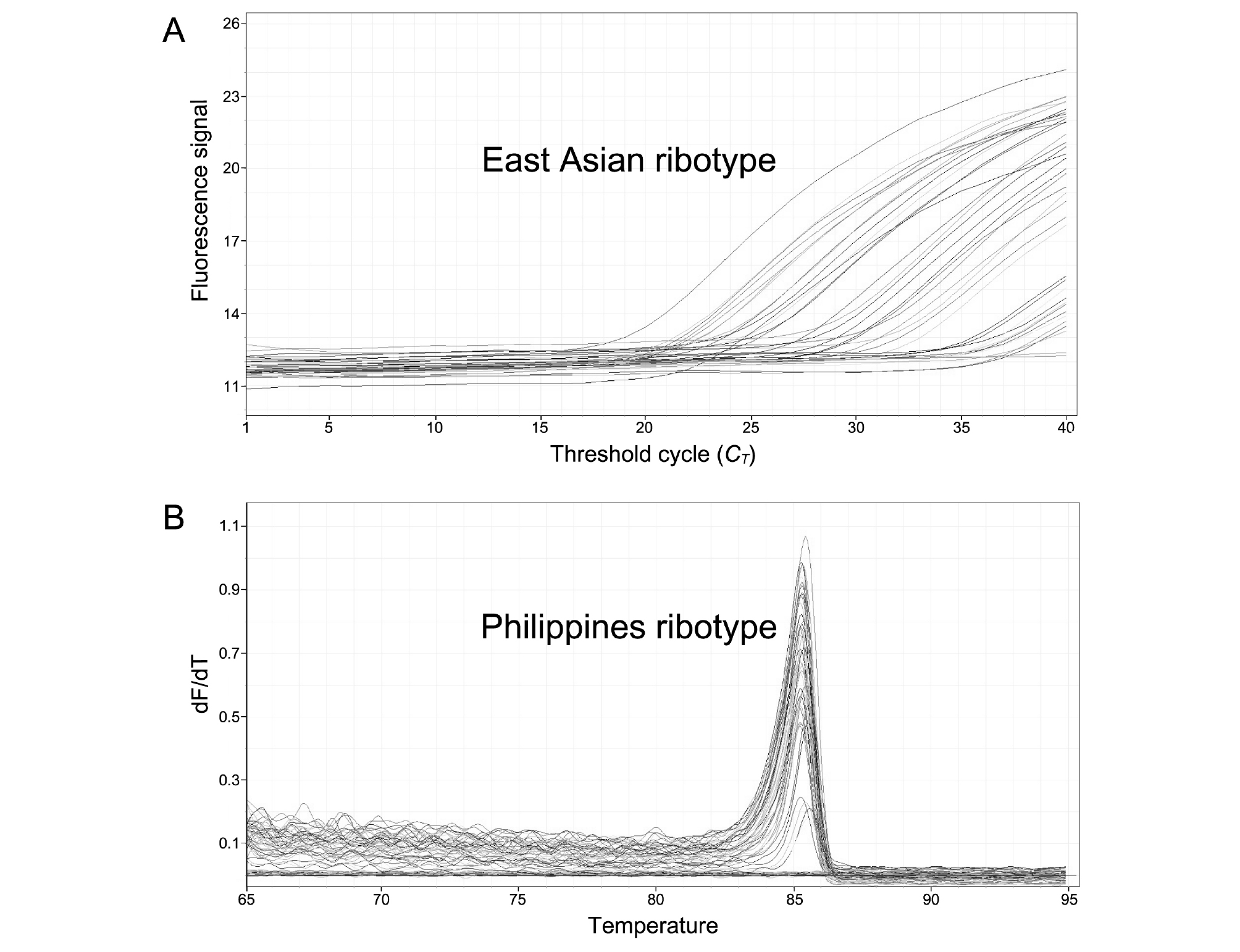
본 연구에서는 qPCR 결과를 양성과 음성 반응으로 표시하였으며, 이를 위해 정량 값이 1 세포 10 L-1 이상은 양성반응 그 미만은 음성반응으로 판정하였다. 검량선 제작은 배양주 세포수를 이용한 방법을 사용하였다. 동아시아타입 C. polykrikoides 배양주 218,400 cells mL-1 (n=1)를 현장시료 처리방법과 동일한 방법으로 DNA를 추출하고 10 배수(10-1∼10-6)로 희석하여 검량선을 제작하였다(R2=0.998). 필리핀 타입의 경우 배양주가 없기 때문에 동아시아 타입 배양주를 이용하여 10 배수(10-1∼10-6)로 희석한 후 검량선을 제작하였다(R2=0.996). 또한 필리핀 타입 분석은 EvaGreen을 사용하는 방법이기 때문에 melting curve 분석결과가 85℃이면서 정량값 1 cell 10 L-1 이상을 양성반응으로 판정하였다(Fig. 2).
3. 결과 및 고찰
qPCR을 이용한 C. polykrikoides 동아시아타입 분석결과 2014년 6∼9월은 월별 양성검출 비율이 70∼100%를 보였다(Table 3). 2015년 6∼9월은 월별 양성검출 비율이 42∼98%를 보였으며, 2016년 6∼8월은 월별 양성검출 비율이 40∼70%를 보였다. 반면, 필리핀타입 분석결과는 2014∼2015년에 불검출이었으며, 2016년 6∼8월은 월별 양성검출 비율이 1∼2%를 보였다. 동아시아 타입은 남해안 전 해역에 분포하였으며, 필리핀 타입은 경남 통영∼전남 고흥의 일부해역에서만 0.0001∼0.002 cells mL-1로 극미량이 검출되었다(Fig. 1, Table 3). 따라서, 동아시아 타입이 남해안의 우점 C. polykrikoides 개체군이며, 필리핀 타입은 매우 소량만 출현함을 알 수 있다.
Table 3. Detection of C. polykrikoides ribotypes using qPCR in the South Sea, Korea during 2014 to 2016. The Philippines ribotype was detected from the four samples at cell densities of 0.0001~0.002 cells mL-1.
2014년과 2015년은 남해안에 대규모 C. polykrikoides 적조가 발생하여 각각 74 억원과 56 억원의 수산피해가 발생하였다. 2014년의 경우 7월 31일∼10월 13일(75 일간) 동안 완도∼삼척 해역에서 최고밀도 20,000 cells mL-1로 발생하였으며, 2015년의 경우 8월 2일∼9월 26일(56 일간) 동안 진도∼울진 해역에서 최고밀도 32,000 cells mL-1로 발생하였다(Table 4). 반면, 2016년은 적조가 매우 소규모로 발생하였으며, 수산피해도 없었다. 2016년 8월 16일∼8월 29일(14 일간) 전남 나로도 해역에서 소규모(최고 밀도 1,500 cells mL-1) 적조띠가 수일간 발생하고 소멸하였다(Table 4). qPCR 동아시아 타입 분석결과도 현장 적조 발생상황과 유사한 양상을 보였는데 2014∼2015년은 8월에 98∼100% 양성검출을 보인 반면, 매우 소규모 적조가 발생한 2016년도는 8월에 70% 양성검출을 보였다. 반면, 필리핀 타입 분석결과의 경우 매우 소규모 적조가 발생한 2016년에만 1∼2% 비율로 양성검출을 보였다.
Table 4. Occurrences of C. polykrikoides blooms and economic losses in Korean waters. Data of C. polykrikoides blooms : NIFS red tide homepage(http://www.nifs.go.kr/redtideInfo)
필리핀 타입은 2009년 8∼10월에도 여수와 남해도 해역에서 출현이 보고 되었다(Park et al., 2014). 2009년과 2016년은 적조발생 규모면에서 유사하였는데 매우 소규모 적조만 발생하였고 수산피해는 없었다. 2009년의 경우 10월 28일∼11월 15일(17일간) 동안 최고 밀도 1,600 cells mL-1로 출현하였으며, 2016년과 유사하게 전남 나로도∼여수 해역에 소규모 적조띠가 수일간 출현하고 소멸하였다(Table 4). 또한 2009년과 2016년의 비슷한 환경요인은 대마난류가 남해안으로 강하게 유입되었다는 공통점이 있다. 2009년과 2016년의 대마난류 세기는 각각 23.7∼31.8 104 km2와 22.0∼25.5 104 km2인 반면 필리핀 타입이 검출되지 않은 2014년과 2015년은 이 보다 낮은 19.0∼23.0 104 km2와 19.0∼20.0 104 km2의 대마난류 세기를 보였다(Table 5). 이는 대마난류 유입이 강할 때 외해역으로부터 필리핀 타입이 남해안으로 유입되었을 가능성을 의미한다. 이전 연구에서도 대마난류를 따라 외해역에서 한국과 일본 연안으로 C. polykrikoides이 유입될 가능성이 제시되었다(Matsuoka et al., 2010). 2007∼2008년에 일본 고토섬 서측(제주도와 일본 큐슈섬 사이)에서 저밀도(0.001∼0.1 cells mL-1)의 C. polykrikoides 유영세포가 현미경하에서 발견되었으며(Matsuoka et al., 2010), 2008년 동중국해에서는 저밀도의 C. polykrikoides (0.001∼0.004 cells mL-1)이 qPCR로 검출되었다(Park et al., 2009). 대마난류는 여름철과 초가을에 강하게 유입되기 때문에 외해역의 유영세포가 이 시기에 남해안으로 유입될 가능성이 있다(Matsuoka et al., 2010 Fig. 5 참고). C. polykrikoides 필리핀 타입이 대마난류 유입경로에서도 검출되었는데 2009년 7월 제주도 동쪽 인근해역에서 qPCR로 검출되었다(Park et al., 2018). Microsatellites 분자기법을 이용하여 대마난류 흐름이 해역별로 서로 다른 C. polykrikoides 유전형 분포에 영향을 줄 수 있다는 보고도 있다(Nagai et al., 2009). 또한 C. polykrikoides 적조띠가 한국 동해안에서 일본 서측해안 방향으로 이동하는 모습이 인공위성으로 관찰됨으로써 C. polykrikoides이 해류를 따라 장거리 이동이 가능하다는 것을 시각적으로 입증한 보고도 있다(Choi et al., 2014; Noh et al., 2018). 따라서 대마난류 유입이 강한시기에 남해안에서 필리핀 타입이 검출된다는 것은 대마난류를 따라 동중국해 등 외해역에서 남해안으로 C. polykrikoides 유영세포가 유입될 수 있음을 본 연구는 보여주고 있다.
Table 5. Comparison between occurrences of C. polykrikoides Philippines ribotype in the South Sea and intensity of Tsushima Warm Current. O : positive detection, X : not detected, ND : no data Data of Tsushima Warm Current area : Japan Meteorological Agency homepage (http://www.data.jma.go.jp/kaiyou/shindan/b_2/kuroshio_flow/kuroshio_flow.html) Data of Philippines ribotype detection : year 2009 (Park et al. 2014), year 2014-2016 (this study)